Tratamiento dirigido. Pruebas rápidas para indicar tratamiento dirigido. Bases para la elección de los antibióticos dirigidos. Criterios para tratamiento combinado en el tratamiento dirigido
CONCEPTO DE TRATAMIENTO DIRIGIDO
PRUEBAS RÁPIDAS PARA INDICAR UN TRATAMIENTO DIRIGIDO
En los pacientes con infecciones graves el tiempo en iniciar un tratamiento adecuado es un factor pronóstico independiente asociado con mortalidad (2). En este sentido, en los pacientes con shock séptico, cada hora que trascurre desde el inicio de la hipotensión hasta la administración de antibióticos activos se traduce en un aumento de mortalidad del 7,6% (3). Por ello resulta obvio que el objetivo a conseguir es acortar el tiempo necesario para llegar a un diagnóstico etiológico y poder así, optimizar el tratamiento antibiótico adecuado en las primeras horas.
A día de hoy, el principal método de diagnóstico etiológico sigue siendo el cultivo directo, que tiene entre otras la importante ventaja de permitir aislar el microorganismo causante de la infección, realizar un antibiograma y si fuera necesario técnicas de tipado molecular. Sin embargo, tiene varios inconvenientes como son la demora diagnóstica, condicionada por el tiempo de incubación necesario para el aislamiento e identificación de cada microorganismo y los falsos negativos, debidos sobre todo al tratamiento antibiótico previo o la presencia de microorganismos exigentes que no crecen en los medios estándares.
Para un diagnóstico precoz etiológico pueden utilizarse diferentes técnicas que incluyen, entre otras, la tinción de Gram, pruebas bioquímicas de identificación fenotípica y la detección de antígenos (orina, plasma), pero a día de hoy las que suscitan mayor interés son los métodos proteómicos y las técnicas moleculares de identificación (4). En la tabla 2 se incluyen algunas de las técnicas más novedosas que pueden emplearse para un diagnóstico rápido en diferentes situaciones clínicas, frecuentes en el entorno de pacientes críticos, como son bacteriemias o fungemias, infecciones relacionadas con catéteres, neumonía, meningitis, infecciones de piel y partes blandas, infecciones del tracto urinario e infecciones por Clostridium difficile.
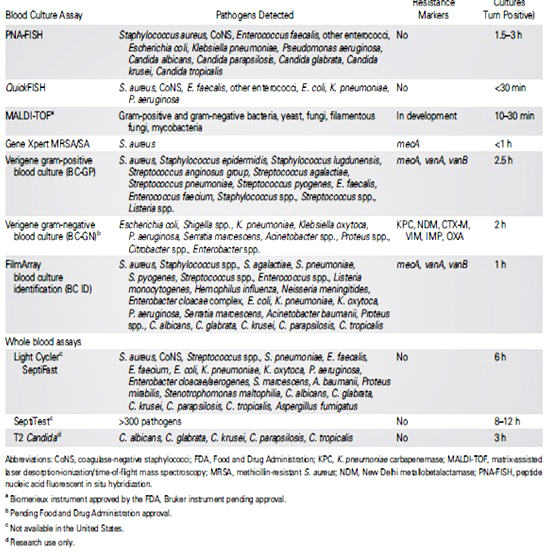
Son técnicas complementarias a los métodos clásicos de identificación pero permiten ofrecer una información de forma más precoz y muchas de ellas aportan además información sobre la sensibilidad del microorganismo porque detectan genes de resistencia antibiótica, resultando de especial utilidad para indicar un tratamiento dirigido o específico de forma precoz.
La espectrofotometría de masas (matrix-assisted laser desorption-ionization time of flight – MALDI-ToF-) es la opción más utilizada en los hospitales en los últimos años. Este método de identificación microbiológica se basa en análisis del espectro proteico generado por un microorganismo, específico para cada género y especie, y su posterior comparación de forma automática con los espectros proteicos de referencia. La generación del perfil proteico se hace mediante espectrofotometría de masas y el esquema del proceso completo se ilustra en la figura 1.
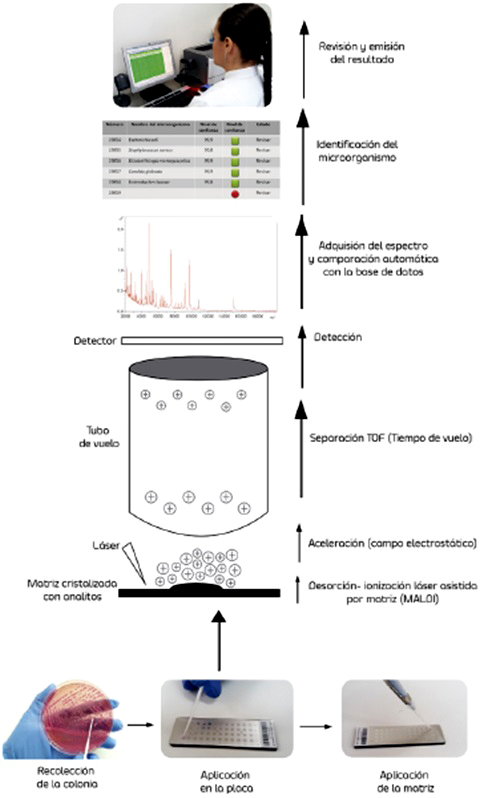
El software proporciona no sólo el resultado de la identificación (ej. E.coli) sino también el grado de fiabilidad de dicha identificación ya que las bases de datos de espectros de referencia forman parte de un sistema patentado y son construidas y mantenidas por los fabricantes. Los sistemas comerciales más empleados que usan la tecnología MALDI-ToF para la identificación de microorganismos son VITEK MS® y MALDI Biotyper®. El resultado final se obtiene en menos de 1h y el nivel de concordancia de esta técnica con los métodos convencionales de identificación de microorganismos supera el 90% en la mayoría de estudios. Su principal aplicación es en hemocultivos y urocultivos, en donde es necesaria una densidad bacteriana superior a 105UFC/mL. En otro tipo de muestras biológicas la posible presencia de otros compuestos orgánicos pueden interferir en el análisis, aunque se ha utilizado también en muestras respiratorias y de LCR.
Dentro de los métodos moleculares, el más utilizado actualmente es el PNA-FISH: técnica de hibridación in situ fluorescente (FISH) que emplea sondas de péptidos de ácidos nucleicos (PNA) marcadas con fluorocromos, las cuales van dirigidas hacia secuencias específicas del ácido ribonucleico ribosomal (ARNr) del microorganismo. Esto permite la identificación rápida (<3h) y específica del agente microbiológico. Desde hace algunos años existe una versión más rápida de esta metodología denominada Quick-FISH que reduce el tiempo de diagnóstico a unos 30min. La sensibilidad y especificidad de ambas técnicas son del 95-100%.
Dentro de las técnicas de amplificación con utilidad clínica a día de hoy destacan: AccuProbe System, que identifica directamente mediante el empleo de una sonda de ADN específica diversos microorganismos Gram +, y GenomEra MRSA/SA, que permite detectar mediante técnicas de PCR aproximadamente en 1h la presencia de bacteriemia por S.aureus al mismo tiempo que su sensibilidad o resistencia a oxacilina.
Además, se están desarrollando nuevas tecnologías que aportan información sobre la sensibilidad a antimicrobianos a partir de un cultivo positivo (FilmArray, Verigene, Gene Xpert, entre otros). Deben ser validadas antes de poder utilizarse en la pràctica, pero que han demostrado gran sensibilidad y especificidad en los estudios clínicos.
En el caso concreto de paciente crítico y teniendo como objetivo acortar el tiempo necesario para llegar al diagnóstico microbiológico y reducir así la mortalidad asociada a la sepsis , se están desarrollando diversas técnicas moleculares para ser aplicadas directamente en la sangre del paciente, sin necesidad de incubación previa y ofreciendo por tanto información de forma muy precoz, Aunque su aplicabilidad clínica debe de concretarse con estudios bien diseñados las más desarrolladas y evaluadas son:
- Light Cyler Septifast: Se basa en la amplificación por PCR a tiempo real de regiones ITS del ADN ribosomal de bacterias y hongos. Detecta 25 especies de bacterias más frecuentes causantes de bacteriemia, 5 especies de Candida y Aspergillus. Los resultados se obtienen en unas 5-6h.
- SepsiTest: sistema semiautomatizado basado en una PCR que utiliza cebadores universales dirigidos a dianas de ARN ribosomal de bacterias y hongos. El resultado de la PCR indica la posible resistencia de bacteriemia o fungemia y, en caso de ser positiva, el producto obtenido se secuencia para obtener la identificación final.
- T2MR: Nuevo sistema sustentado en nanotecnología basado en la resonancia magnética que detecta la presencia de especies del género Candida.
- iDTECT Dx Blood: basado en técnicas de secuenciación de genoma completo. Permite detectar 800 especies bacterianas y 400 virus de muestras de sangre.
- VYOO: emplea una PCR múltiple que permite identificar los microorganismos más frecuentes implicados en una septicemia: 34 bacterias y 7 hongos. Incluye la detección de 5 marcadores de resistencia.
